


※Coming soon!
Ⅲ Glycan Pathway Predictor(GPP)
Glycan Pathway Predictor(GPP) is a tool for dynamically computing the N-glycan biosynthesis pathway from a given glycan structure,
based on the paper by Krambeck et al., 2005, which was later improved in Krambeck et al., 2009.
GPP とは糖転移酵素によるN 型糖鎖の生合成を予測するためのツールです。
基質特異性は数理的モデル[15, 14] で表し、全経路を素早く計算できます。
しかし、この数理的モデルの計算は無限に続く可能性があるため、それを制限するために糖鎖の分子量の最大値をユーザーからの入力によって設定します。
設定した分子量より大きい糖鎖構造は生成しないようになります。
また本モデルは、濃度や局在情報は考慮していないため、論理的な予測を行います。
これに関しては今後、改良する予定です。
利用目的
GPPを用いて理論的に可能な糖鎖構造の全体像がまず見えます。
また、ノックアウト実験などの予測のためにもパスウェイの変化を調べることも可能です。
利用方法
There is one major text field for entering a starting N-glycan structure in KCF format.
This input can also be provided in a text file which can be loaded directly.
A selection menu for the enzymes to work on the generated structures is given.
Multiple enzymes may be selected simultaneously by using the shift- or alt- keys while clicking the enzyme name.
1. Glycan structure information is required in KCF format as input data.
2. Users can write sutructures directly or load a file.
3. Users need to select at least one enzyme from enzyme list.
4. Users need to specify the maximum molecular mass to produce in the pathway.
5. A biosynthetic pathway is predicted by clicking the submit button.
FIGURE5.8 GPP の入力画面。
ユーザーは糖鎖構造の情報をKCF 形式で入力またはファイルをロードし、酵素を選択する。 また、計算に必要な糖鎖の分子量の最大値を設定してsubmit をクリックすることで結果画面を表示させる。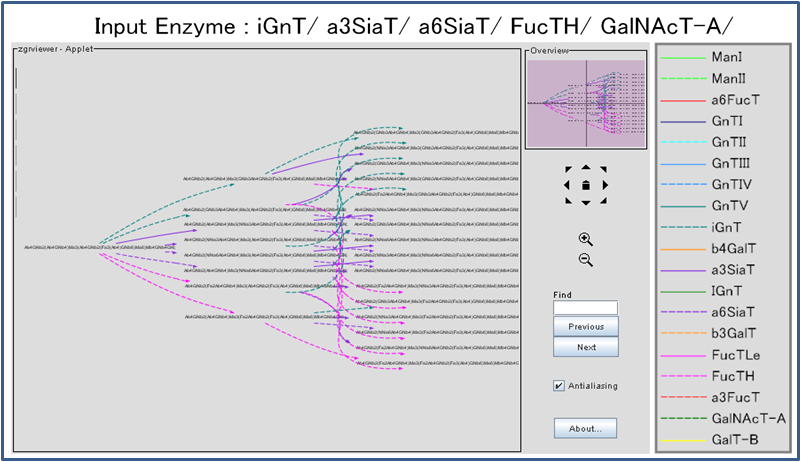
FIGURE5.9 計算された反応経路を示す結果画面。
Each node represents a glycan structure in Linear Code format. A generated pathway is displayed with the ZGRViewer applet so that users can easily see the whole pathway as well as parts of the pathway at the same time.
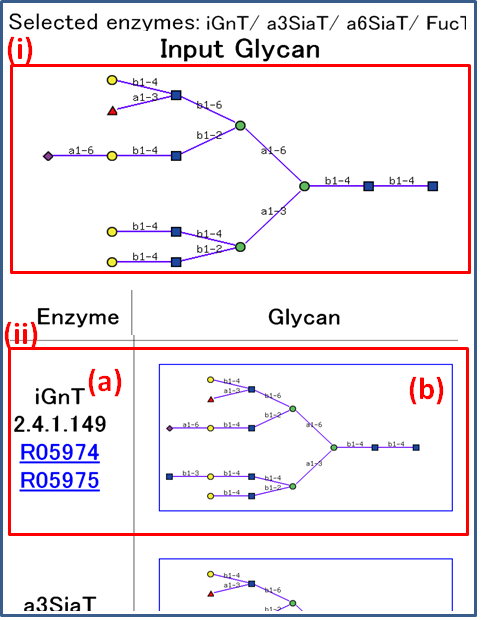
FIGURE5.10 一反応の結果を表示する画面。
Double-clicking on a glycan structure will display the detailed information for the selected structure.
(i) Clicked glycan structure is used as an input glycan.
(ii) Predicted structure modified by enzyme (a) is shown in (b).
Users can see the enzyme information by clicking an Interaction ID that starts with the letter "R".
Clicking on the image of a generated structure (b),
the tool will recalculate the possible enzymes from the original selection that may catalyze a bond on the clicked glycan,
and a new results page will be displayed.
Thus the biosynthesis pathway can be examined one structure at a time.