


※Coming soon!
Ⅳ ProfilePSTMM
Profile PSTMM is a probabilistic model (similar to hidden Markov models, HMMs),
that can find patterns (profiles) in glycan structures that may not necessarily be easily found by the naked eye.
It is implemented based on the work by Aoki-Kinoshita et al., 2006. In particular,
it can predict longer-range patterns in the data,
such as those between the non-reducing and reducing ends.
Thus it is useful for predicting possible recognition patterns in glycans.
ProfilePSTMMとは、機械学習のモデル[9] を用いており、大量の糖鎖構造の情報から共通なプロファイルを抽出することができます。
GlycanMinerとは異なり、共通のプロファイルは部分木に限らず、直接つながっていなくても同時に現れる部分構造が見出されます。
利用目的
ProfilePSTMMの応用例として、糖鎖を認識するタンパク質などが糖鎖のどの部分を認識しているかを予測することができます。
過去に糖鎖アレイデータからレクチンが認識する糖鎖プロファイルを抽出するのに用いたことがあります。
利用方法
Since the profiles that are learned from the data are all stored for easy retrieval,
the tool takes as input a Profile name that is unique.
In the Glycan data section,
glycan structure information can be specified using a list of KEGG Glycan IDs (beginning with the letter 'G' followed by five digits),
one per line, or a list of structures in KCF format.
If a list of IDs is given, an optional parameter for the 'strength' of the particular ID can be specified as a number following each ID.
This may be useful when using Glycan Array data,
for example, when the binding affinity of a particular glycan is stronger or weaker compared to others.
The actual glycan data type specified should be selected,
and the Number of times to shuffle option is used to indicate how many times the learning process should be repeated.
This is useful because this model is a probabilistic model,
whereby local optimums may be found.
In order to ensure that the results contain a global optimum, the learning process should be repeated many times.
Note, however, that increasing this number will also increase computation time.
1. ユーザーは、入力画面でKCF 形式の糖鎖構造情報かGlycan ID を入力またはファイルからロードし、その形式を選択して下さい。
GlycanID を使った場合、オプションとして各ID の右側にタブで区切って数値を指定することもできます。
例として、糖鎖アレイの結合親和性の値を用いることができます。
2. シャッフルする回数を入力して下さい。
ProfilePSTMM は確率モデルの特徴である局所的な最適解を出力することがあるため、複数回実行し、最も高いスコアを最適解として出力します。
3.最後にrun ボタンを押すと解析結果が表示されます。
シャッフル回数が多ければ多いほど、時間はかかりますが、最適解を探すためには多い数が有効です。
FIGURE5.11 ProfilePSTMM の入力画面。
ユーザーは、入力画面でKCF形式の糖鎖構造情報かClycanID を入力またはファイルからロードし、その形式を選択する。 次に、シャッフルする回数を入力する。 そしてrun ボタンを押すと解析結果が表示される。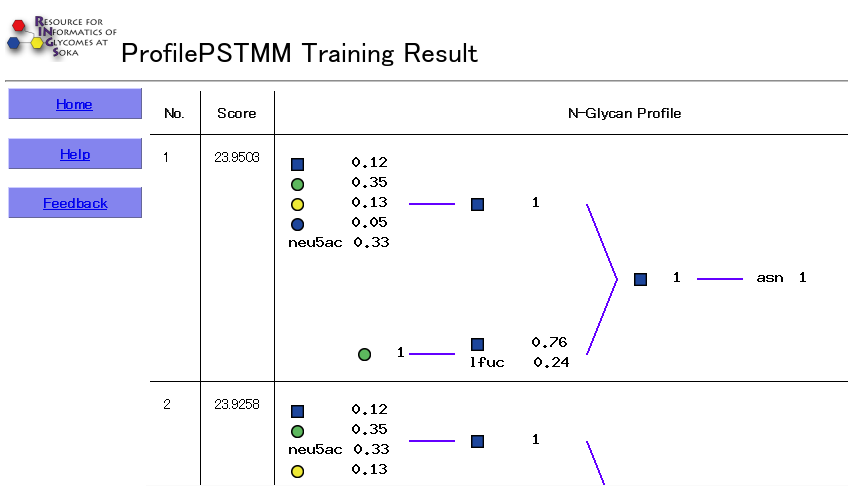
FIGURE5.12 Pro lePSTMM の結果画面。
The results include a list of the learned profiles ranked according to their Likelihood scores, which is computed based on the learned parameters of the probabilistic model. The Glycan Profile is also displayed as a figure, where for each position, the probability of a particular monosaccharide is listed.