


Ⅲ Glycan Pathway Predictor(GPP)
GPP とは糖転移酵素による N 型糖鎖の生合成を予測するためのツールです。
基質特異性は数理的モデル [15, 14] で表し、全経路を素早く計算できます。
しかし、この数理的モデルの計算は無限に続く可能性があるため、それを制限するために糖鎖の分子量の最大値をユーザーからの入力によって設定します。
設定した分子量より大きい糖鎖構造は生成しないようになります。
また本モデルは、濃度や局在情報は考慮していないため、論理的な予測を行います。
これに関しては今後、改良する予定です。
利用目的
GPP を用いて理論的に可能な糖鎖構造の全体像がまず見えます。
また、ノックアウト実験などの予測のためにもパスウェイの変化を調べることも可能です。
利用方法
1. ユーザーは、入力画面で KCF 形式の糖鎖構造情報を入力またはファイルからロードし、酵素の選択と上限となる分子量の最大値を設定して Submit ボタンをクリックして下さい。
2. 結果画面の上部に入力した糖鎖の図と酵素の情報が表示されます。そして入力した酵素に対して選択した酵素が反応しない場合、" No reaction" という文字が表示されます。
反応が行われる場合は Java アプレットを用いて反応経路の全体図が表示されます。
この図は拡大縮小、検索などの機能が利用できます。
3. 各糖鎖には、ハイパーリンクが埋め込んであり一反応の結果を表示させることができます。
図 5.8 GPP の入力画面。
ユーザーは糖鎖構造の情報を KCF 形式で入力またはファイルをロードし、酵素を選択する。 また、計算に必要な糖鎖の分子量の最大値を設定して submit をクリックすることで結果画面を表示させる。
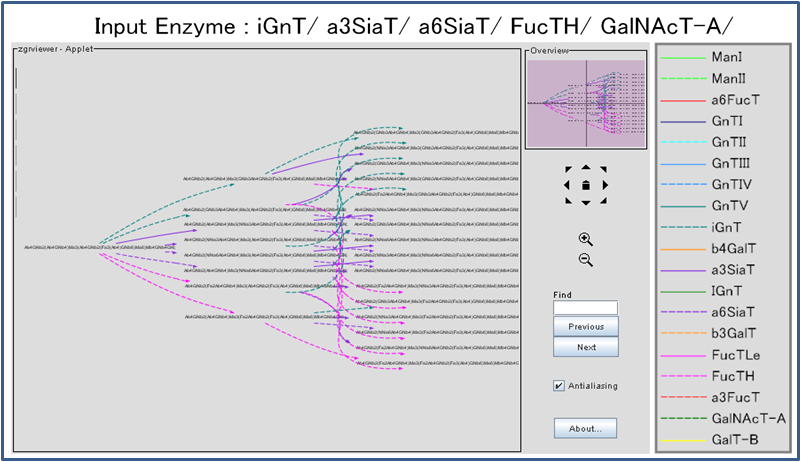
図 5.9 計算された反応経路を示す結果画面。
グラフ中の線は各酵素を表している。 各糖鎖構造はリニアコードで示され、図 5.10 のような一反応の結果画面にリンクしている。
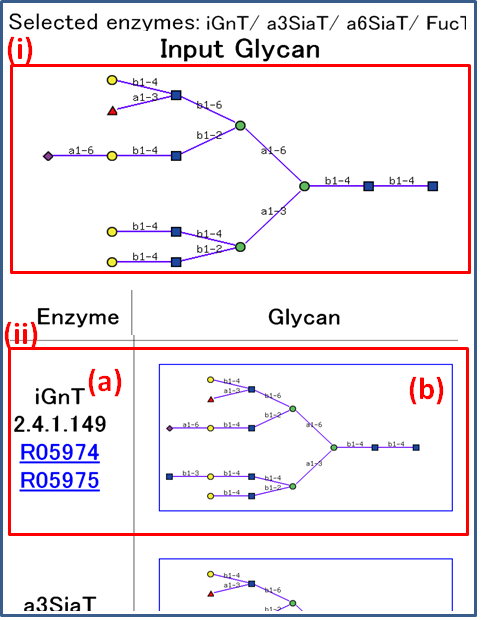
図 5.10 一反応の結果を表示する画面。
(i) ではクリックした糖鎖構造を画像で表示し、(ii) でその糖鎖に対する一段階の反応を表示している。 (ii) の (a) には酵素名とその酵素のEC番号及びリアクションID が表示され、(b) にはその酵素の反応によって生成される糖鎖の構造が画像で表示されている。