


Ⅳ ProfilePSTMM
ProfilePSTMM とは、機械学習のモデル [9] を用いており、大量の糖鎖構造の情報から共通なプロファイルを抽出することができます。
GlycanMiner とは異なり、共通のプロファイルは部分木に限らず、直接つながっていなくても同時に現れる部分構造が見出されます。
利用目的
ProfilePSTMM の応用例として、糖鎖を認識するタンパク質などが糖鎖のどの部分を認識しているかを予測することができます。
過去に糖鎖アレイデータからレクチンが認識する糖鎖プロファイルを抽出するのに用いたことがあります。
利用方法
1. ユーザーは、入力画面で KCF 形式の糖鎖構造情報か Glycan ID を入力またはファイルからロードし、その形式を選択して下さい。
GlycanID を使った場合、オプションとして各 ID の右側にタブで区切って数値を指定することもできます。
例として、糖鎖アレイの結合親和性の値を用いることができます。
2. シャッフルする回数を入力して下さい。
ProfilePSTMM は確率モデルの特徴である局所的な最適解を出力することがあるため、複数回実行し、最も高いスコアを最適解として出力します。
3. 最後に run ボタンを押すと解析結果が表示されます。
シャッフル回数が多ければ多いほど、時間はかかりますが、最適解を探すためには多い数が有効です。
図 5.11 ProfilePSTMM の入力画面。
ユーザーは、入力画面で KCF 形式の糖鎖構造情報か ClycanID を入力またはファイルからロードし、その形式を選択する。 次に、シャッフルする回数を入力する。 そして run ボタンを押すと解析結果が表示される。
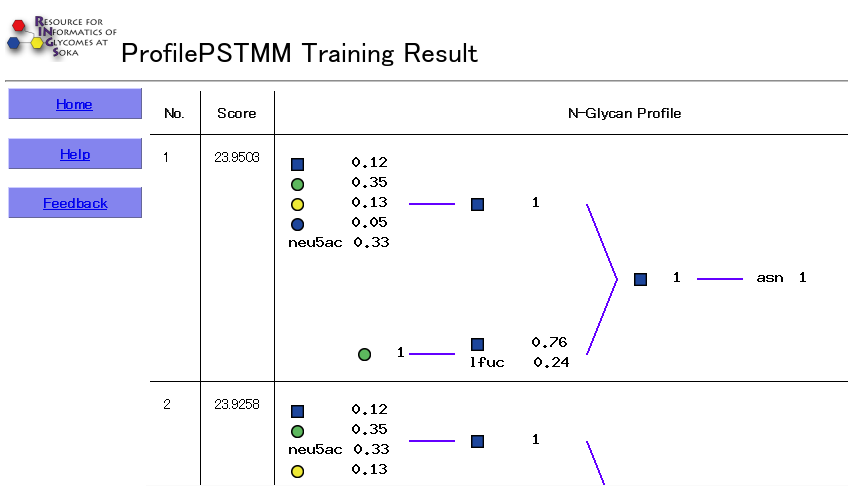
図 5.12 ProfilePSTMM の結果画面。
スコアとプロファイリングの結果が表示される。